Coronaviridae
| Coronaviridae | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Coronaviridae | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Coronaviridae | ||||||||||||||||
| Links | ||||||||||||||||
|
Coronaviridae[3] ist eine Virusfamilie innerhalb der Ordnung Nidovirales. Die Viren innerhalb der Familie werden (fach)umgangssprachlich „Coronaviren“ genannt und gehören zu den RNA-Viren mit den größten Genomen.
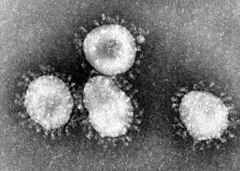
Die ersten Coronaviren wurden bereits Mitte der 1960er-Jahre beschrieben.[4] Als Entdeckerin gilt die britische Virologin June Almeida, der 1966 eine Aufnahme mittels Elektronenmikroskop gelang.[5] Die im elektronenmikroskopischen Bild grob kugelförmigen Viren fallen durch einen Kranz blütenblattartiger Fortsätze auf, die an eine Sonnenkorona erinnern und die ihnen ihren Namen gaben.
Vertreter dieser Virenfamilie verursachen bei allen vier Klassen der Landwirbeltiere (Säugetiere, Vögel, Reptilien, Amphibien) sehr unterschiedliche Erkrankungen. Sie sind genetisch hochvariabel und können so manchmal auch mehrere Arten von Wirten infizieren. Beim Menschen sind sieben Arten von Coronaviren als Erreger von leichten respiratorischen Infektionen (Erkältung / Grippaler Infekt) bis hin zum sog. Schweren akuten Atemwegssyndrom (SARS, Severe acute respiratory syndrome) von Bedeutung.
Unter den menschlichen Coronaviren besonders bekannt geworden sind die folgenden Coronaviren:
- SARS-CoV[-1] (severe acute respiratory syndrome coronavirus [1])
- MERS-CoV (Middle East respiratory syndrome coronavirus)
- SARS-CoV-2 (severe acute respiratory syndrome coronavirus 2)
Sie waren bzw. sind die Auslöser der SARS-Pandemie 2002/2003, der MERS-Epidemie (ab 2012) und der COVID-19-Pandemie (ab 2019).
Anmerkung: Zur klareren und sinnvolleren Unterscheidung zwischen den Coronaviren SARS-CoV und SARS-CoV-2 wird SARS-CoV gelegentlich auch als SARS-CoV-1 bezeichnet.
Merkmale
Erscheinungsbild
Die 60 bis 160 nm großen Viruspartikel (Virionen) besitzen eine Virushülle, in die mehrere verschiedenartige Membranproteine eingelagert sind. Das charakteristische Aussehen der Coronaviren (lateinisch corona ‚Kranz, Krone‘) liegt an vielen etwa 20 nm nach außen vorragenden keulenförmigen Strukturen an der Oberfläche, den Spikes genannten Peplomeren. Sie bestehen aus Anteilen des großen glykosylierten S-Proteins (Spikes-Protein, 180 bis 220 kDa), das hier ein membranverankertes Trimer bildet.[6] Diese Anteile tragen sowohl (S1) die Rezeptor-Bindungs-Domäne (RBD), mit der das Virus an eine Zelle andocken kann,[7] als auch (S2) eine Untereinheit, die als Fusions-Protein (FP) die Verschmelzung von Virushülle und Zellmembran bewirkt.[8]
In geringeren Mengen ist auf der Außenseite das kleinere Envelope-Protein (E-Protein, 9 bis 12 kDa) vorhanden. Nur beim HCoV-OC43 (Humanen Coronavirus OC43) und den Coronaviren der Gruppe 2 (Gattung Betacoronavirus) findet sich zusätzlich das Hämagglutin-Esterase-Protein (HE-Protein, 65 kDa). Das ebenfalls in der Membranhülle verankerte M-Protein (Matrix-Protein, 23 bis 35 kDa) ist dagegen nach innen gerichtet und ein Matrixprotein auf der Innenseite der Virushülle.
Im Inneren der Hülle befindet sich ein vermutlich ikosaedrisches Kapsid, das einen helikalen Nukleoproteinkomplex enthält. Dieser besteht aus dem Nukleoprotein N (50 bis 60 kDa), das mit dem Strang einer einzelsträngigen RNA von positiver Polarität komplexiert ist. Bestimmte Aminosäurereste des N-Proteins interagieren mit dem Matrixprotein M, sodass das Kapsid mit der Membraninnenseite assoziiert ist.
Genom
Das einzelsträngige RNA-Genom der Coronaviren ist etwa 27.600 bis 31.000 Nukleotide (nt) lang, womit Coronaviren die längsten Genome aller bekannten RNA-Viren besitzen.[9]
Am 5'-Ende befinden sich eine 5'-Cap-Struktur und eine nichtcodierende Region (englisch untranslated region, UTR) von etwa 200 bis 400 nt, die eine 65 bis 98 nt kurze, sogenannte Leader-Sequenz enthält. Am 3'-Ende fügt sich eine weitere UTR von 200 bis 500 nt an, die in einem poly(A)-Schwanz endet. Das Genom der Coronaviren enthält 6 bis 14 Offene Leserahmen (englisch open reading frames, ORFs), von denen die beiden größten (die Gene für die Nichtstrukturproteine NSP-1a und 1b) nahe am 5'-Ende liegen und sich mit unterschiedlichen Leserastern etwas überlappen. Die Überlappungsstelle bildet eine Haarnadelstruktur, die bei 20 bis 30 % der Lesedurchläufe einen Leserastersprung bei der Translation an den Ribosomen ermöglicht und so zur Synthese von geringeren Mengen des NSP-1b führt.[10]
Neben der Replikation ihres Genoms synthetisieren die Viren (je nach Virusspezies) 4–9 mRNA-Moleküle, deren 5'- und 3'-Enden mit denen des Genoms identisch sind. Diese "geschachtelten" mRNAs werden auch als "nested set of mRNAs" bezeichnet und haben zur Namensgebung der übergeordneten Virusordnung, Nidovirales (von lateinisch nidus ‚Nest‘), beigetragen.
Im Unterschied zur üblicherweise hohen Fehlerrate der RNA-Polymerase von anderen RNA-Viren, die zu einer Beschränkung der Genomlänge auf etwa 10.000 Nukleotiden führt, wird bei Coronaviren eine relativ hohe genetische Stabilität (Konservierung) unter anderem durch eine 3'-5'-Exoribonuklease-Funktion des Proteins NSP-14 erreicht.[9] Vermutlich bewirkt dieser Korrekturlesemechanismus, dass das antivirale Mittel Ribavirin bei COVID-19 (SARS-CoV-2) nicht wirkt.[11]
Vorkommen und Verbreitung
Bereits 1932 wurde die Infektiöse Bronchitis bei Geflügel untersucht, die vom Infektiöse-Bronchitis-Virus (IBV, Art Avian coronavirus), einem Gammacoronavirus aus der Unterfamilie der Orthocoronavirinae, verursacht wurde. Damals konzentrierten sich die Untersuchungen auf das Krankheitsgeschehen. Aussehen und genetische Verwandtschaftsverhältnisse des Virus waren unbekannt und der Name „Coronaviren“ existierte noch nicht.[12][13]
Die ersten namentlichen Coronaviren wurden Mitte der 1960er Jahre beschrieben.[14][15] Das erstentdeckte Exemplar war das später verlorengegangene humane Coronavirus B814 (nicht klassifiziert[16]). Coronaviren sind genetisch hochvariabel; einzelne Arten aus der Familie Coronaviridae können durch Überwindung der Artenbarriere auch mehrere Arten von Wirten infizieren, also Zoonosen verursachen.
Durch die Überwindung der Artenbarriere sind beim Menschen unter anderem Infektionen mit dem SARS-Coronavirus (SARS-CoV, gelegentlich auch als SARS-CoV-1 bezeichnet) – dem Erreger der SARS-Pandemie 2002/2003 – sowie mit den 2012 neu aufgetretenen Viren der Art Middle East respiratory syndrome coronavirus (MERS-CoV) entstanden.
Die Anfang 2020 von der chinesischen Stadt Wuhan ausgegangene COVID-19-Pandemie wird auf ein bis dahin unbekanntes Coronavirus zurückgeführt, das den Namen SARS-CoV-2 erhielt.[17][18]
Systematik
Etymologie
Der Name „Coronaviren“ – lateinisch corona ‚Kranz, Krone‘ – wurde 1968 eingeführt und hängt mit dem charakteristischen Aussehen dieser Viren unter dem Elektronenmikroskop zusammen. Der erste veröffentlichte Bericht über die Entdeckung gibt die Namensgebung durch die Entdecker wieder als beruhend auf der Ähnlichkeit der Hüllen-Umsäumung der Viren zur Sonnenkorona:
“Particles are more or less rounded in profile; although there is a certain amount of polymorphism, there is also a characteristic “fringe” of projections 200 Å long, which are rounded or petal shaped, rather than sharp or pointed, as in the myxoviruses. This appearance, recalling the solar corona, is shared by mouse hepatitis virus and several viruses recently recovered from man, namely strain B814, 229E and several others.”
„Die Partikel sind mehr oder weniger rundlich im Querschnitt; obwohl es ein gewisses Maß an Polymorphismus gibt, gibt es auch einen charakteristischen „Saum“ aus 200 Å langen Fortsätzen, welche rundlich oder blütenblattförmig sind, statt kantig [?] oder spitz, wie bei den Myxoviren. Dieses an die Sonnenkorona erinnernde Aussehen wird vom Maus-Hepatitis-Virus und einigen kürzlich vom Menschen gewonnenen Viren, namentlich B814, 229E und einigen anderen, geteilt.“
Ein anderer Bericht führt die Wahl der Entdecker auf die Ähnlichkeit der Hüllen-Umsäumung zu einer Krone zurück,[20] beruft sich dabei jedoch auf den Erstbericht, der eine abweichende Beschreibung gibt. Zwei virologische Referenzwerke enthalten ein Kapitel, bei dem ein Autor namensgleich mit einer Person aus der o. g. Entdeckergruppe ist („Kenneth McIntosh“ vs. „K. McIntosh“), und, in dem jeweils die „Kronen-Etymologie“ gegeben wird.[21][22]
Strukturierung
Die Familie Coronaviridae wird auf der Grundlage von phylogenetischen Eigenschaften in zwei Unterfamilien und aktuell fünf Gattungen eingeteilt (eine sechste ist vorgeschlagen).[2] Eigenschaften wie Wirtsspektrum, Organspektrum oder Erkrankungsart spielen bei der Klassifikation keine Rolle.[23]
Die aktuellen beiden Unterfamilien heißen Orthocoronavirinae und Letovirinae. Dabei ist Orthocoronavirinae die weitaus größere von beiden. Letovirinae ist ein deutlich jüngeres Taxon innerhalb der Familie Coronaviridae, und bisher ist nur eine einzige Letovirenart bekannt: Microhyla letovirus 1.[24]
Die fünf aktuellen Gattungen heißen: Alphacoronavirus, Betacoronavirus, Deltacoronavirus, Gammacoronavirus und Alphaletovirus. Eine weitere Gattung „Epsiloncoronavirus“[25] könnte noch dazukommen.[2] Die früheren Gattungen Torovirus und Bafinivirus finden sich heute in der Unterfamilie Torovirinae in der Nidoviren-Familie Tobaniviridae.[24][2]
Die Anzahl der Arten ändert sich laufend, insbesondere seit der SARS-Pandemie 2002/2003. Seitdem hatte sich die Forschungstätigkeit im Bereich der Coronaviren massiv verstärkt. Die Arten sind in eine größere Zahl Untergattungen eingeordnet. Diese sollen bei der systematischen Einordnung noch nicht eingehend beschriebener bzw. zukünftig entdeckter Coronavirusarten helfen und sind in der systematischen Übersicht (siehe unten) aufgelistet.[23]
Innere Systematik
Angegeben sind nur die wichtigsten Spezies:
- Familie Coronaviridae
- Unterfamilie Letovirinae
- Gattung Alphaletovirus
- Untergattung Milecovirus
- Spezies Microhyla letovirus 1 (MLeV)[26] (*)
- Untergattung Milecovirus
- Gattung Alphaletovirus
- Unterfamilie Orthocoronavirinae, ehem. Unterfamilie Coronavirinae,[27] ehem. Gattung Coronavirus[28]
- Gattung Alphacoronavirus, ehem. Phylogruppe Gruppe-1-Coronaviren[28]
- Untergattung Colacovirus
- Spezies Bat coronavirus CDPHE15
- Untergattung Decacovirus
- Spezies Rhinolophus ferrumequinum alphacoronavirus HuB-2013
- Untergattung Duvinacovirus
- Spezies Human coronavirus 229E (dt. Humanes Coronavirus 229E, HCoV-229E, kommt auch in Fledermäusen vor.[11])
- Untergattung Luchacovirus
- Spezies Lucheng Rn rat coronavirus
- Untergattung Minacovirus
- Spezies Ferret coronavirus
- Spezies Mink coronavirus 1
- Untergattung Minunacovirus
- Spezies Miniopterus bat coronavirus 1
- Spezies Miniopterus bat coronavirus HKU8 (Bat-CoV-HKU8)
- Untergattung Myotacovirus
- Spezies Myotis ricketti alphacoronavirus Sax-2011
- Spezies Nyctalus velutinus alphacoronavirus SC-2013
- Untergattung Pedacovirus
- Spezies Felines infectious peritonitis virus (FIPV) (Alte Einordnung – siehe bei Felines Coronavirus (FCoV))
- Spezies Porcine epidemic diarrhea virus (dt. Porzines Epidemische-Diarrhoe-Virus, PEDV)[29][30][31]
- Spezies Scotophilus bat coronavirus 512
- Untergattung Rhinacovirus
- Spezies Rhinolophus bat coronavirus HKU2 (Bat-CoV-HKU2)
- Untergattung Setracovirus
- Spezies Human coronavirus NL63 (HCov-NL63, kommt auch in Fledermäusen vor.[11])
- Spezies NL63-related bat coronavirus strain BtKYNL63-9b[34]
- Untergattung Soracovirus
- Untergattung Sunacovirus
- Untergattung Tegacovirus
- Spezies Alphacoronavirus 1 (*)
- Subspezies Canines Coronavirus (englisch canine coronavirus, CCoV), ehem. Spezies Canine coronavirus[28]
- Subspezies Felines Coronavirus (englisch feline coronavirus, FCoV, Jetzt: Felines Infektiöses Peritonitis-Virus, englisch feline infectious peritonitis virus, FIPV), ehem. Spezies Feline coronavirus[28]
- Subspezies Transmissible-Gastroenteritis-Virus (TGEV), ehem. Spezies Transmissible gastroenteritis virus[28][35] (infiziert auch Schweine)[30]
- Subspezies Porcine respiratory coronavirus (PRCV)[30][36]
- Spezies Alphacoronavirus 1 (*)
- Untergattung Colacovirus
- Gattung Betacoronavirus, ehem. Phylogruppe Gruppe-2-Coronaviren[28]
- Untergattung Embecovirus
- Spezies Betacoronavirus 1
- Subspezies Bovines Coronavirus (BCoV)
- Subspezies Equines Coronavirus (ECoV-NC99)
- Subspezies Humanes Coronavirus OC43 (HCoV-OC43, befällt auch Schimpansen,[37] kommt auch in Nagetieren vor.[11])
- Subspezies Porzines hämagglutinierendes Enzephalomyelitis-Virus (HEV, PHEV)[30][38]
- Subspezies Humanes Enterisches Coronavirus (HECoV)[28]
- Spezies China Rattus coronavirus HKU24 (RtCov-HKU24)
- Spezies Human coronavirus HKU1 (HCoV-HKU1, kommt auch in Nagetieren vor[11])
- Spezies Murine coronavirus (*)
- Subspezies Murines Hepatitis-Virus (englisch mouse hepatitis virus, MHV), ehem. Spezies Murine hepatitis virus[28]
- Subspezies Ratten-Coronavirus (englisch rat coronavirus, RtCoV),[39] ehem. Spezies Rat coronavirus[28]
- Subspezies puffinosis coronavirus (PV), bei Schwarzschnabel-Sturmtauchern (Puffinus puffinus), ehem. Spezies Puffinosis coronavirus[28]
- Spezies Betacoronavirus 1
- Untergattung Hibecovirus
- Spezies Bat Hp-betacoronavirus Zhejiang2013
- Untergattung Merbecovirus
- Spezies Hedgehog coronavirus 1
- Spezies Middle East respiratory syndrome-related coronavirus (dt. MERS-Coronavirus, MERS-CoV)
- Spezies Pipistrellus bat coronavirus HKU5 (Bat-CoV-HKU5)
- Spezies Tylonycteris bat coronavirus HKU4 (Bat-CoV-HKU4)
- Untergattung Nobecovirus
- Spezies Rousettus bat coronavirus GCCDC1
- Spezies Rousettus bat coronavirus HKU9 (Bat-CoV-HKU9)
- Untergattung Sarbecovirus
- Spezies Severe acute respiratory syndrome-related coronavirus (dt. SARS-assoziiertes Coronavirus, englisch SARS-related coronavirus, SARS-CoV, SARSr-CoV), ehem. Spezies Severe acute respiratory syndrome coronavirus (bis 2009, englisch SARS coronavirus, dt. SARS-Coronavirus, namensidentisch mit damals einziger Subspezies)[40]
- Subspezies Severe acute respiratory syndrome coronavirus (SARS-CoV, SARS-Coronavirus, auch SARS-CoV-1), Erreger von SARS[41]
- Subspezies Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2, auch[42][43] englisch 2019-novel Coronavirus, 2019-nCoV oder Wuhan seafood market pneumonia virus), Erreger von COVID-19
- Stamm SARS-CoV/SARS-CoV-2 RdRp (künstliche Mutante: ursprüngliches SARS-Virus SARS-CoV-1 mit ausgetauschtem RdRp-Gen von SARS-CoV-2)[44][45]
- Stamm Rhinolophus affinis bat coronavirus RaTG13 (BatCoV-RaTG13, Bat_SL-CoV_RaTG13), gefunden in Java-Hufeisennasen (Rhinolophus affinis, englisch intermediate horseshoe bat) in der chinesischen Provinz Yunnan[46][47][48][49] (mit Gensequenz KP876546 zu BtCoV/4991 Polymerase)[50]
- Stamm Bat coronavirus Rc-o319 (BatCoV-Rc-o319), gefunden im Kot von Horn-Hufeisennasen[51] (Rhinolophus cornutus, englisch little Japanese horseshoe bat) in Japan[52]
- Stamm Bat coronavirus RmYN01 (BatCoV-RmYN01 BetaCoV/Rm/Yunnan/YN01/2019)[53]
- Stamm Rhinolophus malayanus bat coronavirus RmYN02 (BatCoV-RmYN02 BetaCoV/Rm/Yunnan/YN02/2019),[53][54] gefunden in Malaiischen Hufeisennasen (R. malayanus).
- Stämme SARS-ähnliche Fledermaus-Coronaviren ohne Zuordnung:[49] Bat_SL-CoV_ZC45 (bat-SL-CoVZC45),[50] Bat_SL-CoV_ZXC21 (bat-SL-CoVZXC21),[50] Bat_SL-CoV_Rs4231, Bat_SL-CoV_Rs4247, Bat_SL-CoV_Rs7327, Bat_SL-CoV_Rs9401, Bat_SL-CoV_Rf9402, Bat_SL-CoV_WIV16, Bat_SL-CoV_GX2013, Bat_SL-CoV_Anlong-112, Bat_SL-CoV_Shaanxi2011, Bat_SL-CoV_Yunnan2011, Bat_SL-CoV_HuB2013, Bat_SL-CoV_Longguan, Bat_SL-CoV_As6526, Bat_SL-CoV_YN2013, Bat_SL-CoV_YN2018B, Bat_SL-CoV_YN2018C, Bat_SL-CoV_HKU3-3, Bat_SL-CoV_HKU3-7, Bat_SL-CoV_HKU3-12, Bat_SL-CoV_279, Bat_SL-CoV_SC2018, Bat_SL-CoV_BM48-31, Bat_SL-CoV_BtKY72, Bat_SL-CoV_YNLF-34C, Bt-SLCoV Rp3 (infiziert Rhinolophus sinicus),[55] SARS-CoV SZ3 und SZ16 (infizieren Larvenroller).[56]
- vorgeschl. Spezies Pangolin coronavirus[57] (Manis-CoV[58][42] oder englisch pangolin SARS-like coronavirus (Pan_SL-CoV),[59] Pangolin-CoV[53])
- Klade Pan_SL-CoV_GX gefunden in Malaiischen Schuppentieren (Manis javanica, englisch Sunda pangolin, vom chinesischen Zoll in der Provinz Guangxi beschlagnahmt)[49]
- Stamm Pan_SL-CoV_GX/P4L
- Stamm Pan_SL-CoV_GX/P2V
- Stamm Pan_SL-CoV_GX/P1E
- Stamm Pan_SL-CoV_GX/P5L
- Stamm Pan_SL-CoV_GX/P5E
- Stamm Pan_SL-CoV_GX/P3B
- Klade Pan_SL-CoV_GD gefunden in Malaiischen Schuppentieren (Manis javanica, englisch Sunda pangolin, vom chinesischen Zoll in der Provinz Guangdong beschlagnahmt)[49]
- Metagenom MP789 (Schuppentier),[50]
- Klade Pan_SL-CoV_GX gefunden in Malaiischen Schuppentieren (Manis javanica, englisch Sunda pangolin, vom chinesischen Zoll in der Provinz Guangxi beschlagnahmt)[49]
- Spezies Severe acute respiratory syndrome-related coronavirus (dt. SARS-assoziiertes Coronavirus, englisch SARS-related coronavirus, SARS-CoV, SARSr-CoV), ehem. Spezies Severe acute respiratory syndrome coronavirus (bis 2009, englisch SARS coronavirus, dt. SARS-Coronavirus, namensidentisch mit damals einziger Subspezies)[40]
- Untergattung Embecovirus
- Gattung Gammacoronavirus, ehem. Phylogruppe Gruppe-3-Coronaviren[28]
- Untergattung Brangacovirus
- Spezies Goose coronavirus CB17
- Untergattung Cegacovirus
- Spezies Beluga whale coronavirus SW1 (BWCoV-SW1)
- Untergattung Igacovirus
- Spezies Avian coronavirus (dt. Vogel-Coronavirus) (*)
- Subspezies Truthahn-Coronavirus (TCoV)
- Subspezies Fasanen-Coronavirus (PhCoV)
- Subspezies Infektiöse-Bronchitis-Virus (englisch avian infectious bronchitis virus, IBV), Erreger der Infektiösen Bronchitis bei Vögeln
- Spezies Avian coronavirus (dt. Vogel-Coronavirus) (*)
- Untergattung Brangacovirus
- Gattung Deltacoronavirus
- Untergattung Andecovirus
- Spezies Wigeon coronavirus HKU20 (WiCoV-HKU20)
- Untergattung Buldecovirus (inkl. ehem Untergattung Moordecovirus[66])
- Spezies Bulbul coronavirus HKU11 (BuCoV-HKU11) (*)
- Spezies Coronavirus HKU15
- Spezies Munia coronavirus HKU13 (dt. Bronzemännchen-Coronavirus HKU13, MunCoV HKU13)
- Spezies Common moorhen coronavirus HKU21 (CMCoV_HKU21,[68] zuvor in ehem Untergattung Moordecovirus[66])
- Spezies White-eye coronavirus HKU16
- Spezies Thrush coronavirus HKU12 (dt. Drossel-Coronavirus HKU12, ThCoV-HKU12)[69][70]
- vorgeschl. Spezies Sparrow coronavirus HKU17 (dt. Sperlings-Coronavirus HKU17, SpCoV-HKU17)[71][72]
- Untergattung Herdecovirus
- Spezies Night heron coronavirus HKU19
- Untergattung Andecovirus
- vorgeschl. Gattung „Epsiloncoronavirus“[25]
- vorgeschl. Untergattung „Tropepcovirus“[25]
- vorgeschl. Spezies „Tropidophorus coronavirus 118981“[25] (Tsin-CoV 118981,[25] auch „Guangdong chinese water skink coronavirus“[73][74]), bei chinesischen Wasserskinken (Tropidophorus sinicus)[73] (*)
- vorgeschl. Untergattung „Tropepcovirus“[25]
- Gattung Alphacoronavirus, ehem. Phylogruppe Gruppe-1-Coronaviren[28]
Unterfamilie Torovirinae (inklusive Gattungen Torovirus und Bafinivirus)→ Familie Tobaniviridae[28]
Im folgenden Kladogramm nach Mang Shi et al. (2016) wurden die Bezeichnungen gemäß ICTV MSL #35 (Stand März 2020) aktualisiert:[72][2]
| Coronaviridae |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Äußere Systematik
Ordnung Unterordnung Familie Nidovirales Abnidovirineae Abyssoviridae Arnidovirineae Arteriviridae Cremegaviridae Gresnaviridae Olifoviridae Cornidovirineae Coronaviridae Mesnidovirineae Medioniviridae Mesoniviridae Monidovirineae Mononiviridae Nanidovirineae Nanghoshaviridae Nanhypoviridae Ronidovirineae Euroniviridae Roniviridae Tornidovirineae Tobaniviridae
Taxonomische Hintergründe
Unterfamilien
Bis 2018 bestand Coronaviridae aus den Unterfamilien Coronavirinae und Torovirinae. Davor war sie bigenerisch, bestehend aus den Gattungen (Genera) Coronavirus und Torovirus.[75][24]
2009, im Zuge der Weiterentwicklung der Familie, war die Gattung Coronavirus zur Unterfamilie Coronavirinae erhoben worden. Die Unterfamilie enthielt dieselben Viren wie zuvor die Gattung. Sie war neben die neue Unterfamilie Torovirinae gestellt worden, die unter anderem die Gattung Torovirus enthielt.[75]
2018 wurde die Unterfamilie Coronavirinae in Orthocoronavirinae umbenannt. Näheres im Abschnitt Orthocoronavirinae.[24]
Gattungen
Die Viren innerhalb der alten Gattung Coronavirus waren auf der Grundlage von phylogenetischen und serologischen Eigenschaften der Arten in drei nicht-taxonomische, monophyletische Gruppen (Kladen) unterteilt worden, die man früher auch als HCoV-229E-ähnliche (Gruppe 1), HCoV-OC43-ähnliche (Gruppe 2) und IBV-ähnliche (Gruppe 3) Coronaviren bezeichnet hatte. Gruppe 2 wurde weiter in die vier ebenfalls monophyletischen Untergruppen 2A bis 2D unterschieden.[76][77][78]
Während der 2009 stattfindenden Bildung der neuen Unterfamilie Coronavirinae aus der alten Gattung Coronavirus wurden die damaligen drei informellen, aber lange und gut etablierten monophyletischen Gruppen zu den heutigen Gattungen Alpha- bis Gammacoronavirus (in der Reihenfolge des griechischen Alphabetes: Alpha, Beta, Gamma, Delta, Epsilon, …).[76][75] Gattung Deltacoronavirus[79] kam später dazu, eine weitere Gattung „Epsiloncoronavirus“[25] könnte bald dazukommen. Die Gattung Torovirus blieb hingegen unter diesem Namen bestehen und wurde in die neue Unterfamilie Torovirinae eingeordnet. Dieser wurde neben Torovirus noch die neue Gattung Bafinivirus von bazilliformen (stäbchenförmigen) Fischviren hinzugefügt.[75]
2018 verschwanden die Toroviren dann gänzlich aus der Familie Coronaviridae, gleichzeitig kamen die Letoviren neu hinzu.[24]
Mit „Toroviren“ sind hier alle Viren der Unterfamilie Torovirinae in der damaligen Form gemeint. Diese Unterfamilie umfasste die Viren der Gattungen Toro- und Bafinivirus und weitere Viren. Diese Viren wurden 2018 in die neue Nidoviren-Familie Tobaniviridae mit einer neugestalteten Unterfamilienstruktur gestellt, wodurch unter anderem Namenszweideutigkeiten endeten: Nun sind wieder nur die Viren der Gattung Torovirus auch als Toroviren klassifiziert.[24]
| Gattungen / gattungsähnliche Gruppen | Familienzugehörigkeit | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| bis 2009 | ab 2009 | ab 2011 | ab 2018 | kommend (Stand Juli 2020) | bis 2018 | ab 2018 | ||||
| Coronavirus |
|
|
Alphacoronavirus | Coronaviridae | Coronaviridae | |||||
|
|
Betacoronavirus | ||||||||
|
|
Gammacoronavirus | ||||||||
| Deltacoronavirus | ||||||||||
| „Epsiloncoronavirus“ | (n. a.) | |||||||||
| Alphaletovirus | ||||||||||
| Torovirus | Torovirus | Coronaviridae | Tobaniviridae | |||||||
| Bafinivirus | ||||||||||
Legende: gattungsähnlich gebrauchte, monophyletische Gruppen Unterfamilien
Untergattungen
2018 wurden zum ersten Male auch eine ganze Reihe Untergattungen in der Familie Coronaviridae definiert. Darunter auch Untergattungen in der Gattung Betacoronavirus (siehe ebenda). So wie zuvor diese Gattung aus der bekannten Phylogruppe 2 gebildet worden war, wurden auch die nun unter den Namen Linie A bis Linie D bekannten Untergruppen 2A bis 2D in Untergattungen umgemünzt.[76][24]
2018 wurden zum ersten Male auch eine ganze Reihe Untergattungen in der Familie Coronaviridae definiert. Der Name der Untergattungen entspricht einem Schema sprechender Namen in Form von Neologismen. Sie sind durchweg Kofferworte (nach ICTV-Code[80] ein sogenanntes siglum (englisch/lateinisch)) wie z. B. Sarbecovirus entsprechend SARS-like betacoronavirus.
Ein ausdrücklicher Grund dafür war ein Klassifikationsstau durch viele noch nicht eingehend beschriebene und daher noch einzuordnende Viren. Auch waren viele Neuentdeckungen und Neubeschreibungen durch die großen Fortschritte in der Genomanyalyse und die verstärkte Forschungstätigkeit im Bereich der Coronaviren seit der SARS-Pandemie 2002/2003 zu erwarten. Man wollte durch diesen Aspekt der taxonomischen Struktur einen rationellen Rahmen für die systematische Einordnung bieten, der nur noch auf die genomischen Eigenschaften der Viren abstellt. Denn die waren schon lange als der einzig relevante Gesichtspunkt für die Einordnung festgelegt worden.[23]
Unter den Untergattungen sind auch solche in der Gattung Betacoronavirus (siehe ebenda). So wie zuvor diese Gattung aus der bekannten Phylogruppe 2 gebildet worden war, wurden auch die nun unter den Namen Linie A bis Linie D bekannten Untergruppen 2A bis 2D in Untergattungen umgemünzt.[76][24]
Bedeutung
Medizin
Erkrankungen
Coronaviren verursachen bei verschiedenen Wirbeltieren wie Säugetieren, Vögeln, Fischen und Fröschen sehr unterschiedliche Erkrankungen.
Beim Menschen sind diverse Arten des Coronavirus als Erreger von leichten respiratorischen Infektionen (Erkältungskrankheiten) bis hin zum schweren akuten Atemwegssyndrom von Bedeutung. Eine ursächliche Beteiligung an Gastroenteritiden ist möglich, spielt jedoch klinisch und zahlenmäßig keine große Rolle.[81] Insgesamt sind sieben humanpathogene Coronaviren bekannt (Stand Februar 2020): neben SARS-CoV(-1), SARS-CoV-2 und MERS-CoV noch HCoV-HKU1, HCoV-OC43 (alle zur Gattung Betacoronavirus), HCoV-NL63 und HCoV-229E (beide zur Gattung Alphacoronavirus). Die vier letztgenannten verursachen etwa 5–30 % aller akuten respiratorischen Erkrankungen und führen typischerweise zu Rhinitis, Konjunktivitis, Pharyngitis, gelegentlich zu einer Laryngitis oder einer Mittelohrentzündung (Otitis media). Auch Infektionen des unteren Respirationstraktes sind möglich, insbesondere bei Koinfektionen mit anderen respiratorischen Erregern (wie Rhinoviren, Enteroviren, Respiratory-Syncytial-Viren (RSV), Parainfluenzaviren). Schwere Krankheitsverläufe werden vor allem bei vorbestehenden Erkrankungen, insbesondere des kardiopulmonalen Systems, beobachtet und im Zusammenhang mit Transplantationen (Immunsuppression);[81] im Normalfall treten nur vergleichsweise geringfügige Symptome auf.[41]
Bei einer Verlaufsstudie über acht Jahre – vor dem Ausbruch von COVID-19 – in ausgesuchten Haushalten mit etwa tausend Personen in Michigan, USA, waren knapp 1000 akute Atemwegserkrankungen durch HCoV-Infektionen verursacht (zumeist OC43). Auffällig war das saisonal begrenzte Auftreten dieser Infektionen in den Monaten Dezember bis April/Mai, was aber nicht notwendig bei SARS-CoV-2 genauso vorausgesetzt werden kann.[82][83][84]
Unterfamilien
Die Unterfamilie entstand 2018 aus der Umbenennung der Unterfamilie Coronavirinae. Diese wiederum entstand, indem die Gattung Coronavirus 2009 zur Unterfamilie erhoben wurde (und die Endung -virus in -virinae geändert wurde).[75][24]
Parallel bestand bis 2018 innerhalb der Familie Coronaviridae jeweils ein Toroviren-Schwestertaxon mit analoger Benennung: Torovirus als Schwestergattung von Coronavirus und Torovirinae als Schwesterunterfamilie von Coronavirinae.[75][24] Dadurch ergab sich die Situation, dass Toroviren namentlich sowohl Coronaviren waren, wegen ihrer Zugehörigkeit zur Familie Coronaviridae, als auch Nicht-Coronaviren, wegen ihrer Nicht-Zugehörigkeit zur Unterfamilie / Untergattung Coronavirinae / Coronavirus. So etwas war nie ungewöhnlich in der Virentaxonomie. Ähnliche Verwicklungen bestanden zeitweise zwischen Toroviren und Bafiniviren.
Die Toroviren entsprachen zudem nicht oder nur sehr eingeschränkt dem Namen „Coronavirus“.
Hinter dem Namen „Corona-virus“ steht die Idee von einer sonnenartigen Grundgestalt umgeben von einer sonnenartigen Korona. Damit ist ein in elektronenmikroskopischen Bildern als scheibenförmig erscheinendes Virus gemeint, das von einem deutlichen, abgesetzten Kranz aus keulen- oder blütenblattförmigen Fortsätzen eingerahmt wird. (Siehe auch Abschnitt Etymologie) Aber weder die Toro- noch die Bafiniviren waren scheiben- bzw. kugelförmig. (Obschon Toroviren dazu gebracht worden seien sollen, in Zellkultur zum Teil kugelförmige Virionen zu produzieren.[85]) Sie hatten ihre Namen gerade daher, dass sie Torus- bzw. Bazillus-förmig waren, also Ring- bzw. Stäbchen-förmig. Die Toroviren waren tatsächlich sogar stäbchenförmige Gebilde, die zu einem fast geschlossenen Ring oder einer mondsichelförmigen Gestalt gebogen waren.[76]
Zu den Unterschieden gesellten sich dann noch Abweichungen in der Genomstruktur zwischen Toro- und Bafiniviren auf der einen Seite und Coronaviren auf der anderen Seite. Das Genom kodierte z. B. nicht das Hüllenprotein E der anderen Coronaviren und war insgesamt sehr viel einfacher organisiert. Insbesondere fehlten fast alle typischen Hilfsgene der anderen Coronaviren.[76]
Toro- und Bafiniviren hatten zudem tubuläre, helikale Nukleokapside, während die der anderen Coronaviren lose gewunden waren. Auch waren die Kapsidproteine weniger als halb so groß wie die der anderen Coronaviren. Letztlich wichen die Bafiniviren auch noch im Wirtsspektrum (= aquatisch) von den Viren innerhalb Coronavirinae / Coronavirus (= terrestrisch) ab.[76]

Aufgrund dieser Unterschiede wurden bis 2018 die Viren der damaligen Unterfamilie Coronavirinae bzw. der vormaligen Gattung Coronavirus typischerweise als „echte Coronaviren“ bezeichnet. So grenzte man sie von den bloß formalen Coronaviren aus der Gruppe der Toro- und Bafiniviren ab. Dann fand 2018 die Entfernung der Toro- und Bafiniviren aus der Familie Coronaviridae statt. Gleichzeitig wurde eine Namensänderung von Coronavirinae zur heutigen Unterfamilie Orthocoronavirinae vorgenommen.[76][75] Seitdem steht der wohlabgegrenzte Name „Orthocoronaviren“ für die Viren dieser Gruppe zur Verfügung.
Formal gesehen bezeichnet der Name „Coronaviren“ nun alle Viren der Familie Coronaviridae und – soweit alleinstehend – sonst nichts. Das entspricht auch den ICTV-Regeln der Virus-Taxonomie, der Mehrdeutigkeiten bei der (Neu-)Benennung von Taxa verbietet.[80]:3.16
- Sog. „echte“ Coronaviren
Severe acute respiratory syndrome-related coronavirus (hier SARS-CoV-2)
Avian coronavirus (hier IBV, zu Gammacoronavirus)
Betacoronavirus 1 (hier HCoV-OC43)
Middle East respiratory syndrome-related coronavirus (MERS-CoV)
- Bis 2018 ebenfalls Coronaviren
Typus von Torovirus (hier BEV, Art Equine torovirus)
Typus von Bafinivirus (hier WBV, Art White bream virus)
Da die im Rahmen der Namensänderung hinzugekommenen Letoviren den Orthocoronaviren deutlich ähnlicher sind, ist der Name „Coronaviren“ nun auch ohne weiteres zutreffend für alle Viren der Familie Coronaviridae und es müsste keine Mehrdeutigkeiten mehr geben.[80]:3.16[26] Trotzdem werden weiterhin in erster Linie die Viren der Unterfamilie Orthocoronavirinae als die Coronaviren bezeichnet.[86] Das mag an der noch geringen Erforschtheit der Gruppe der Letoviren (Unterfamilie Letovirinae) liegen.
Letovirinae
Die Unterfamilie wurde 2018 aufgrund der Entdeckung der Froschvirusart Microhyla letovirus 1 geschaffen.
Von den Letoviren als Gruppe ist noch nicht viel bekannt, da sie bisher nur durch diese eine noch nicht sehr eingehend erforschte Art vertreten werden. Sie stimmen in allen wesentlichen Eigenschaften mit den Orthocoronaviren überein, bilden aber in statistischen Verwandtschaftsanalysen der Genome eine unabhängige Gruppe, die zu weit entfernt von den Orthocoronaviren ist, um etwa innerhalb der Unterfamilie Orthocoronavirinae eine neue Gattung neben den anderen dort vorhandenen Gattungen zu bilden.[26][24]
Eine mögliche nahe Verwandte von Microhyla letovirus 1 ist die vorgeschlagene und bisher unklassifizierte Nidovirenart „Pacific salmon nidovirus“[87][88] (PsNV). Es ist wahrscheinlich, dass sie innerhalb der Familie Coronaviridae zu einer eigenen Gattung parallel zu Alphaletovirus gehören wird. Ob innerhalb derselben Unterfamilie Letovirinae (also ein weiteres Letovirus), oder jenseits davon ist noch unklar.[89]
Literatur
- David M. Knipe, Peter M. Howley (eds.-in-chief): Fields’ Virology. 5. Auflage. 2 Bände, Lippincott Williams & Wilkins, Philadelphia 2007, ISBN 0-7817-6060-7.
- C. M. Fauquet, M. A. Mayo et al. Eighth Report of the International Committee on Taxonomy of Viruses. Elsevier Academic Press, London/ San Diego 2005, ISBN 0-12-249951-4.
- A. M. Q. King, M. J. Adams, E. B. Carstens, E. J. Lefkowitz (Hrsg.): Virus Taxonomy. Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier, Amsterdam u. a. 2012, ISBN 978-0-12-384684-6, S. 806–828.
- S. Modrow, D. Falke, U. Truyen: Molekulare Virologie. Spektrum-Lehrbuch, 2. Auflage, Akademischer Verlag, Heidelberg/Berlin 2003, ISBN 3-8274-1086-X, S. 214–226.
- P. S. Masters: The molecular biology of coronaviruses. In: Advances in virus research. Band 66, 2006, S. 193–292, doi:10.1016/S0065-3527(06)66005-3 (freier Volltextzugriff). PMID 16877062.
Siehe auch
- June Almeida machte von Coronaviren erste Bilder mit einem Elektronenmikroskop
Weblinks
- Wie gefährlich ist das neuartige Corona-Virus? – Hamburger Abendblatt vom 5. Februar 2020.
- Alex Knapp: The secret history of the first coronavirus 229E, in Forbes vom 12. April 2020 — Forschungsgeschichte der humanpathogenen Coronaviren (229E, OC43, HKU1, NL63, IBV)
- Jürgen Schönstein: Lesetipp: Drosten-Porträt in science, auf: scienceblogs.de — dito: Zeitschiene
Einzelnachweise
- ↑ ICTV Master Species List 2018b v2. MSL #34v, März 2019.
- ↑ a b c d e f g h ICTV: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus. EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ Jens H. Kuhn, Peter B. Jahrling: Clarification and guidance on the proper usage of virus and virus species names. In: Archives of Virology. Band 155, April, S. 445–453, 4. März 2010, Tabelle 1, S. 12, Ze 1, Sp 3, doi:10.1007/s00705-010-0600-9, PMID 20204430, PMC 2878132 (freier Volltext) – (englisch, Volltext [PDF; 64 kB; abgerufen am 18. Juni 2020]): “Taxa refer to theoretical averages of groups of viruses; hence taxa are not preceded by articles.”
- ↑ Faktencheck zu Coronavirusmythen – Von Chlordioxid, Vertuschung und Zwiebeln. auf: n-tv.de vom 18. März 2020. Quelle: RKI
- ↑ Denise Gellene: Overlooked No More: June Almeida, Scientist Who Identified the First Coronavirus. Auf: nytimes.com vom 8. Mai 2020 (Update 10. Mai 2020); abgerufen am 5. August 2020.
- ↑ B. Delmas, H. Laude: Assembly of Coronavirus Spike Protein Into Trimers and Its Role in Epitope Expression. In: Journal of Virology. Band 64, Nr. 11, November 1990. PMID 2170676, PMC 248586 (freier Volltext), S. 5367–5375.
- ↑ Structure of "Spike" Protein in New Coronavirus Determined. Auf: ChemistryViews.org vom 21. Februar 2020; abgerufen am 3. März 2020.
- ↑ Spike Protein / S Protein. auf Sino Biological.com; abgerufen am 3, März 2020.
- ↑ a b M. R. Denison, R. L. Graham, E. F. Donaldson, L. D. Eckerle, R. S. Baric: Coronaviruses: an RNA proofreading machine regulates replication fidelity and diversity. In: RNA Biology. Band 8, Nr. 2, März-April 2011, S. 270–279, doi:10.4161/rna.8.2.15013, ISSN 1555-8584. PMID 21593585, PMC 3127101 (freier Volltext).
- ↑ P. C. Y. Woo, M. Wang, S. K. P. Lau, H. Xu, R. W. S. Poon, R. Guo, B. H. L. Wong, K. Gao, H.-w. Tsoi, Y. Huang, K. S. M. Li, C. S. F. Lam, K.-h. Chan, B.-j. Zheng, K.-y. Yuen: Comparative Analysis of Twelve Genomes of Three Novel Group 2c and Group 2d Coronaviruses Reveals Unique Group and Subgroup Features. In: Journal of Virology. Band 81, Nr. 4, Februar 2007, S. 1574–1585, doi:10.1128/JVI.02182-06, PMID 17121802, PMC 1797546 (freier Volltext).
- ↑ a b c d e David Cyranoski: Virologie: Porträt eines Killers. Online-Ausgabe des Artikels in Spektrum der Wissenschaft. Nr. 8, August 2020, S. 40–49.
- ↑ C. B. Hudson, F. R. Beaudette: INFECTION OF THE CLOACA WITH THE VIRUS OF INFECTIOUS BRONCHITIS. In: Science. Band 76, Nr. 1958. American Association for the Advancement of Science, 8. Juli 1932, ISSN 0036-8075, S. 34, doi:10.1126/science.76.1958.34-a, PMID 17732084 (englisch, Volltext [abgerufen am 10. August 2020]).
- ↑ Yassine Kasmi, Khadija Khataby, Amal Souiri, Moulay Mustapha Ennaji: Coronaviridae: 100,000 Years of Emergence and Reemergence. Buchkapitel. In: Moulay Mustapha Ennaji (Hrsg.): Emerging and Reemerging Viral Pathogens. Band 1: Fundamental and Basic Virology Aspects of Human, Animal and Plant Pathogens. Elsevier/Academic Press, 2020, ISBN 978-0-12-819400-3, S. 127–149, doi:10.1016/B978-0-12-819400-3.00007-7 (englisch, Volltext [PDF; 2,6 MB; abgerufen am 10. August 2020]).
- ↑ Faktencheck zu Coronavirusmythen – Von Chlordioxid, Vertuschung und Zwiebeln. auf: n-tv.de vom 18. März 2020. Quelle: RKI
- ↑
Jeffrey Kahn, Kenneth McIntosh: History and Recent Advances in Coronavirus Discovery. In: The Pediatric Infectious Disease Journal. Band 24, Nr. 11, 2005, S. S223–S227, doi:10.1097/01.inf.0000188166.17324.60.
National Foundation for Infectious Diseases: Coronaviruses. Auf: nfid.org; eingesehen am 23. März 2020. - ↑ Stuart Siddell, Steven Myint: Coronaviruses. In: Steven Myint, D. A. J. Tyrrell, David Taylor-Robinson (Hrsg.): Viral and Other Infections of the Human Respiratory Tract. Chapman & Hall, London 1996, ISBN 978-94-011-7932-4 (Volltext als PDF).
- ↑ New-type coronavirus causes pneumonia in Wuhan: expert. Auf: xinhuanet.com vom 9. Januar 2020.
WHO: Pneumonia of unknown cause – China. Disease Outbreak News vom 5. Januar 2020. - ↑ Hamburger Abendblatt – Hamburg: Wie gefährlich ist das neuartige Corona-Virus? 5. Februar 2020, abgerufen am 18. März 2020 (deutsch).
- ↑ Virology: Coronaviruses. In: Nature. Band 220, 16. November 1968, ISSN 1476-4687, S. 650, doi:10.1038/220650b0 (englisch, Volltext [PDF; 1,8 MB; abgerufen am 18. Juni 2020] Erstmeldung über Entdeckung der Coronaviren).
- ↑ A. F. Bradburne: Antigenic Relationships amongst Coronaviruses. In: Archiv für Virusforschung. Band 31. Springer-Verlag, 6. Februar 1970, S. 352–364, doi:10.1007/BF01253769 (englisch, Volltext [PDF; 923 kB; abgerufen am 18. Juni 2020]).
- ↑ Kenneth McIntosh (für dieses Kapitel): Feigin and Cherry's Textbook of Pediatric Infectious Disease. 6. Auflage. Saunders / Elsevier, Philadelphia, PA (USA) 2009, ISBN 978-1-4160-4044-6, section XVII, subsection 10, chapter 189A. Coronaviruses and Toroviruses, S. 2380 (englisch, Volltext des Kapitels [PDF; 265 kB; abgerufen am 7. Juli 2020]): “[T]he genus Coronavirus of the family Coronaviridae, named for the crown-like appearance of their surface projections on electron microscopy.”
- ↑ Kenneth McIntosh, Stanley Perlman (beide für dieses Kapitel): Mandell, Douglas, and Bennett's Principles and Practice of Infectious Diseases. 8. Auflage. Band 2. Saunders / Elsevier, Philadelphia, PA (USA) 2014, ISBN 978-1-4557-4801-3, chapter 157. Coronaviruses, Including Severe Acute Respiratory Syndrome (SARS) and Middle East Respiratory Syndrome (MERS), S. 1928, rechte Spalte (englisch, 3904 S., Volltext des Kapitels [PDF; 1,5 MB; abgerufen am 8. Juli 2020]): “[T]he name coronavirus (the prefix corona denoting the crownlike appearance of the surface projections) was chosen to signify this new genus.”
- ↑ a b c
- Raoul J. de Groot, John Ziebuhr, Leo L. Poon, Patrick C.Woo, Pierre Talbot, Peter J.M. Rottier, Kathryn V. Holmes, Ralph Baric, Stanley Perlman, Luis Enjuanes, Alexander E. Gorbalenya: Revision of the family Coronaviridae. Proposal. In: Virus Taxonomy. History. Revision 2009, Juni. International Committee on Taxonomy of Viruses (ICTV), 2008, Proposal-Code 2008.085-126V (englisch, ictvonline.org [PDF; 175 kB; abgerufen am 5. Mai 2020]): “The naming of coronavirus genera is according to the Greek alphabet. The viruses grouped in currently recognized genera form distinct monophylogenetic clusters, but do not share other obvious traits (host tropism, organ tropism, type of disease) to suggest a common denominator. Hence, the CSG proposes to use a neutral nomenclature, […].”
- Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Kap. „Coronaviridae“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.): „Viruses that share more than 90 % aa sequence identity in the conserved replicase domains are considered to belong to the same species. This 90 % identity threshold serves as the sole species demarcation criterion.“
- Proposal zu ICTV-Revision № 2018a: 2017.013S. (PDF in ZIP-Ordner; 4,49 MB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch): „We would like to stress that we expect the proposed taxonomy structure to provide a framework for the rationalization of the molecular and biological properties of viruses in these two families, which, in many cases, remain to be determined and, therefore, cannot be used to evaluate the validity of the proposed structure.“
- ↑ a b c d e f g h i j k J. Ziebuhr, R.S. Baric, S. Baker, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: 2017.012_015S.A.v1.Nidovirales. In: Virus Taxonomy. History. Revision 2018a, Juli. International Committee on Taxonomy of Viruses (ICTV), 18. Februar 2017, Proposal-Code 2017.012_015S (ictvonline.org [ZIP; 5,1 MB; abgerufen am 7. Mai 2020]).
- ↑ a b c d e f J. Ziebuhr, S. Baker, R.S. Baric, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: 2019.021S. Proposal-Dokument in ZIP. In: International Committee on Taxonomy of Viruses (Hrsg.): Virus Taxonomy. History. Revision 2019, Dokument-Name in ZIP: 2019.021S.A.v1.Corona_Tobaniviridae.docx. EC 51, Berlin April 2019, Proposal-Code 2019.021S (ictvonline.org [ZIP; 1,8 MB; abgerufen am 9. Juni 2020] ratifiziert im März 2020, noch nicht vollständig veröffentlicht, Proposal-Webseite auf ictvonline.org).
- ↑ a b c Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the _Coronavirinae_, the proposed genus Alphaletovirus. In: Virology. Band 524. Elsevier Inc., November 2018, S. 160–171, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Proposal zu ICTV-Revision № 2018a: 2017.013S. (PDF in ZIP-Ordner; 4,49 MB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- ↑ a b c d e f g h i j k l ICTV: Revision of the family Coronaviridae. Taxonomic proposal to the ICTV Executive Committee 2008.085-126V.
- ↑ New coronavirus emerges from bats in China, devastates young swine. Auf: ScienceDaily. 4. April 2018.
- ↑ a b c d e f g Caitlin E. Edwards et al. Swine acute diarrhea syndrome coronavirus replication in primary human cells reveals potential susceptibility to infection, in: PNAS, 12. Oktober 2020, doi:10.1073/pnas.2001046117
- ↑ NCBI: Porcine epidemic diarrhea virus (species)
- ↑ Zhou et al: Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. In: Nature research, life sciences reporting summary, Letter. Juni 2017, doi:10.1038/s41586-018-0010-9.
- ↑ Nadja Podbregar: Schweine-Coronavirus: Droht ein Artsprung? – Neues Virus SADS-CoV kann sich auch in menschlichen Zellen vermehren, auf: scinexx.de vom 14. Oktober 2020.
- ↑ ICTV: ICTV Taxonomy history: NL63-related bat coronavirus strain BtKYNL63-9b. EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ Turmeric could have antiviral properties. Auf: eurekalert.org vom 17. Juli 2020, Quelle: Microbiology Society.
- ↑ NCBI: Porcine respiratory coronavirus (no rank)
- ↑ Nadja Podbregar: Coronavirus: Sind auch Menschenaffen gefährdet?. Auf: scinexx.de vom 30. März 2020.
- ↑ NCBI: Porcine hemagglutinating encephalomyelitis virus (no rank)
- ↑ NCBI: Rat coronavirus. (no rank)
- ↑ Ben Hu, Lei-Ping Zeng, Xing-Lou Yang, Xing-Yi Ge, Wei Zhang, Bei Li, Jia-Zheng Xie, Xu-Rui Shen, Yun-Zhi Zhang, Ning Wang, Dong-Sheng Luo, Xiao-Shuang Zheng, Mei-Niang Wang, Peter Daszak, Lin-Fa Wang, Jie Cui, Zheng-Li Shi; Christian Drosten (Hrsg.): Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. In: PLOS Pathogens. 30. November 2017, doi:10.1371/journal.ppat.1006698.
- ↑ a b Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The Proximal Origin of SARS-CoV-2. Auf: virologica.org, Quelle: ARTIC Network, 17. Februar 2020.
- ↑ a b Chengxin Zhang, Wei Zheng, Xiaoqiang Huang, Eric W. Bell, Xiaogen Zhou, and Yang Zhang: Protein Structure and Sequence Reanalysis of 2019-nCoV Genome Refutes Snakes as Its Intermediate Host and the Unique Similarity between Its Spike Protein Insertions and HIV-1. In: Journal of Proteome Research. Band 19, Nr. 4. American Chemical Society, 22. März 2020, S. 1351–1360, doi:10.1021/acs.jproteome.0c00129 (englisch, Volltext [PDF; 7,1 MB; abgerufen am 6. Juni 2020] Halb kursiv gesetzt: Manis-CoV, scheinbar bzgl. Wirtstier-Typografie: Manis javanica): “2019-nCoV and the Manis coronavirus (Manis-CoV)”
- ↑ a b Tommy Tsan-Yuk Lam, Marcus Ho-Hin Shum, Hua-Chen Zhu, Yi-Gang Tong, Xue-Bing Ni, Yun-Shi Liao, Wei Wei, William Yiu-Man Cheung, Wen-Juan Li, Lian-Feng Li, Gabriel M. Leung, Edward C. Holmes, Yan-Ling Hu, Yi Guan: Identification of 2019-nCoV related coronaviruses 1 in Malayan pangolins in southern China. auf: bioRxiv vom 18. Februar 2020, doi:10.1101/2020.02.13.945485 (Preprint), doi:10.1038/s41586-020-2169-0 (neuere Version: „Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins“)
- ↑ Andrea J. Pruijssers et al. Remdesivir inhibits SARS-CoV-2 in human lung cells and chimeric SARS-CoV expressing the SARS-CoV-2 RNA polymerase in mice. In: Cell Reports. 7. Juli 2020, doi:10.1016/j.celrep.2020.107940 (freies Pre-Proof)
- ↑ Tina Hesman Saey: Remdesivir may work even better against COVID-19 than we thought. Auf: ScienceNews vom 13. Juli 2020.
- ↑ a b c Alexandre Hassanin: Coronavirus Could Be a 'Chimera' of Two Different Viruses, Genome Analysis Suggests. Auf: sciencealert vom 24. März 2020 (Quelle: THE CONVERSATION).
- ↑ Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The Proximal Origin of SARS-CoV-2. In: virological.org, Quelle: ARTIC Network, 17. Februar 2020, Nature
- ↑ NCBI: Bat coronavirus RaTG13.
- ↑ a b c d Xiaojun Li, Elena E. Giorgi, Manukumar Honnayakanahalli Marichannegowda, Brian Foley, Chuan Xiao, Xiang-Peng Kong, Yue Chen, S. Gnanakaran, Bette Korber, Feng Gao: Emergence of SARS-CoV-2 through recombination and strong purifying selection. In: Science Advances, AAAs. 29. Mai 2020, eabb9153, doi:10.1126/sciadv.abb9153
- ↑ a b c d Jose Halloy, Erwan Sallard, José Halloy, Didier Casane, Etienne Decroly, Jacques van Helden: Tracing the origins of SARS-COV-2 in coronavirus phylogenies, in: HAL vom 16. Juli 2020, HAL Id: hal-02891455 (Preprint)
- ↑ Hufeisennasenfledermäuse. Schutzgemeinschaft Deutscher Wald, Oberursel vom 16. Dezember 2015.
- ↑ Smriti Mallapaty: Coronaviren in Japan und Kambodscha eng verwandt mit Pandemievirus, auf: Spektrum.de vom 6. Dezember 2020.
- ↑ a b c Hong Zhou, Xing Chen, Tao Hu, Juan Li, Hao Song, Yanran Liu, Peihan Wang, Di Liu, Jing Yang, Edward C.Holmes, Alice C.Hughes, Yuhai Bi, Weifeng Shi: A novel bat coronavirus closely related to SARS-CoV-2 contains natural insertions at the S1/S2 cleavage site of the spike protein. In: Current Biology. 11. Mai 2020 (Pre-proof), doi:10.1016/j.cub.2020.05.023.
- ↑ Jacinta Bowler: Researchers Find Another Virus in Bats That's Closely Related to SARS-CoV-2. Auf: sciencealert Vom 12. Mai 2020.
- ↑ Dezhong Xu, Huimin Sun, Haixia Su, Lei Zhang; Jingxia Zhang, Bo Wang, Rui Xu: SARS coronavirus without reservoir originated from an unnatural evolution, experienced the reverse evolution, and finally disappeared in the world. In: Chinese Medical Journal. Band 127, Nr. 13, 5. Juli 2014, S. 2537–2542, doi:10.3760/cma.j.issn.0366-6999.20131328
- ↑ Ming Wang et al. SARS-CoV Infection in a Restaurant from Palm Civet. In: Emerging Infectious Diseases journal. (Emerg Infect Dis.) Band 11, Nr. 12, Dezember 2005, S. 1860–1865, doi:10.3201/eid1112.041293, PMC PMC3367621 (freier Volltext). PMID 16485471
- ↑ NCBI: Pangolin coronavirus. (species)
- ↑ Samantha Black: Scientists use bioinformatics to investigate origin of SARS-CoV-2. (PDF) In: The Science Advisory Board. 27. März 2020, abgerufen am 6. Juni 2020 (englisch, Halb kursiv gesetzt: Manis-CoV, scheinbar bzgl. Wirtstier-Typografie: Manis javanica): „[…] the researchers drafted a genome for Manis-CoV that was used in comparison to SARS-CoV-2 using metagenomic samples.“
- ↑ Xiaojun Li, Elena E. Giorgi, Manukumar Honnayakanahalli Marichannegowda, Brian Foley, Chuan Xiao, Xiang-Peng Kong, Yue Chen, S. Gnanakaran, Bette Korber, Feng Gao: Emergence of SARS-CoV-2 through recombination and strong purifying selection. In: Science Advances – AAAs. Artikel-Nr. abb9153. American Association for the Advancement of Science, 29. Mai 2020, ISSN 2375-2548, doi:10.1126/sciadv.abb9153 (englisch, Volltext [PDF; 3,1 MB; abgerufen am 7. Juni 2020]): “These pangolin SARS-like CoVs (Pan_SL-CoV) form two distinct clades corresponding to their locations of origin: the first clade, Pan_SL-CoV_GD, sampled from Guangdong (GD) province in China, […] the second clade, Pan_SL-CoV_GX, sampled from Guangxi (GX) province […].”
- ↑ NCBI Sequence Read Archive (SRA).
- ↑ NCBI: Virome of dead pangolin individuals: SRR10168376.
- ↑ a b c Chengxin Zhang et al. Protein Structure and Sequence Reanalysis of 2019-nCoV Genome Refutes Snakes as Its Intermediate Host and the Unique Similarity between Its Spike Protein Insertions and HIV-1. In: American Chemical Society: J. Proteome Res. 22. März 2020, doi:10.1021/acs.jproteome.0c00129; PrePrint, PrePrint Volltext (PDF) vom 8. Februar 2020.
- ↑ NCBI: Virome of dead pangolin individuals: SRR10168377.
- ↑ a b Xingguang Li, Junjie Zai, Qiang Zhao, Qing Nie, Yi Li, Brian T. Foley, Antoine Chaillon: Evolutionary history, potential intermediate animal host, and cross‐species analyses of SARS‐CoV‐2. In: Journal of Medical Virology. Band 27. Februar 2020, doi:10.1002/jmv.25731, PDF. PMID 32104911, researchgate
- ↑ NCBI: Virome of dead pangolin individuals: SRR10168378.
- ↑ a b
Datenbankeintrag und relevantes Proposal:
- Eintrag: ICTV Taxonomy history: Moordecovirus. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 9. Juni 2020 (englisch).
- Proposal 2019.021S zu ICTV-Revision № 2019: Create eight new species in the subfamily Orthocoronavirinae of the family Coronaviridae and four new species and a new genus in the subfamily Serpentovirinae of the family Tobaniviridae. (Eine von mehreren PDFen in ZIP-Ordner; 725 kB) 2019.021S. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 9. Juni 2020 (englisch).
- ↑ NCBI: Porcine coronavirus HKU15 (no rank)
- ↑ Spreadsheet 2017.012-015S zu ICTV-Revision № 2018a: 2017.012-015S. (Eine XLSX-Datei in ZIP-Ordner; 4,49 MB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 9. Juni 2020 (englisch).
- ↑ Sander van Boheemen: Virus Discovery and Characterization using Next-Generation Sequencing. Proefschrift Erasmus Universiteit Rotterdam, Rotterdam 2014, ISBN 978-90-8891-932-9, Figur 3.
- ↑ NCBI: Thrush coronavirus HKU12. (species)
- ↑ NCBI: Sparrow coronavirus HKU17. (species)
- ↑ a b Mang Shi,Xiabn-Dan Lin, Jun-Huia Tian et al. Redefining the invertebrate RNA virosphere. In: Nature 540, S. 539–543, 23. November 2016, doi:10.1038/nature20167, via ResearchGate (Volltext), Supplement (PDF).
- ↑ a b Strain Details for Guangdong chinese water skink coronavirus Strain ZGLXR118981. In: ViPR-Homepage. 2. Mai 2020, abgerufen am 16. Juni 2020 (englisch).
- ↑ NCBI: Guangdong chinese water skink coronavirus (species)
- ↑ a b c d e f g Raoul J. de Groot, John Ziebuhr, Leo L. Poon, Patrick C.Woo, Pierre Talbot, Peter J.M. Rottier, Kathryn V. Holmes, Ralph Baric, Stanley Perlman, Luis Enjuanes, Alexander E. Gorbalenya: Revision of the family Coronaviridae. Proposal. In: Virus Taxonomy. History. Revision 2009, Juni. International Committee on Taxonomy of Viruses (ICTV), 2008, Proposal-Code 2008.085-126V (englisch, ictvonline.org [PDF; 175 kB; abgerufen am 5. Mai 2020]).
- ↑ a b c d e f g h Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Kap. „Coronaviridae“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Kenneth McIntosh (für dieses Kapitel): Feigin and Cherry's Textbook of Pediatric Infectious Disease. 6. Auflage. Saunders / Elsevier, Philadelphia, PA (USA) 2009, ISBN 978-1-4160-4044-6, section XVII, subsection 10, chapter 189A. Coronaviruses and Toroviruses, S. 2380 (englisch, Volltext des Kapitels [PDF; 265 kB; abgerufen am 7. Juli 2020] Zusätzlich zum Zitat ein- bis mehrmalige Verwendung im Kapitel (einschließlich Quellenangaben) von: »HCoV-229E-like isolates«, »HCoV-OC43-like isolates«, »HCoV-229E-like viruses«, »HCoV-229E-like strains«, »HCoV-OC43-like strains«, »229E-like coronavirus«, »“avian infectious bronchitis virus-like” viruses (coronaviruses)«, »“IBV-like” virus«, »“IBV-like” viruses«.): “When sufficient virus has been recovered for characterization, most isolates have proved to be similar, either HCoV-229E–like or HCoV-OC43–like,57 although several HCoVs, including the very first isolate HCoV-B814, remain antigenically uncharacterized. [… nächste Spalte …] The antigenic interrelationships of these four proteins have permitted arrangement of both the animal coronaviruses and HCoVs into three groups. The two known HCoV serotypes, each along with several other mammalian coronaviruses, have been placed in group I (HCoV-229E) or II (HCoV-OC43), and avian infectious bronchitis virus is the single member of group III.”
- ↑ Francesca Rovida, Elena Percivalle, Maurizio Zavattoni, Maria Torsellini, Antonella Sarasini, Giulia Campanini, Stefania Paolucci, Fausto Baldanti, M. Grazia Revello, Giuseppe Gerna: Monoclonal antibodies versus reverse transcription‐PCR for detection of respiratory viruses in a patient population with respiratory tract infections admitted to hospital. In: Journal of Medical Virology. Band 75, Ausgabe 2, Februar 2005, S. 336-347. Wiley-Liss, 15. Dezember 2004, S. 336, linke Spalte, doi:10.1002/jmv.20276, PMID 15602736, PMC 7166428 (freier Volltext) – (englisch, Volltext [PDF; 278 kB; abgerufen am 8. Juli 2020]): “[H]uman coronavirus (hCoV) groups I (229E‐like) and II (OC43‐like) […] were searched for by RT‐PCR […].”
- ↑
Datenbankeintrag und zugehöriger Revisionsvorschlag:
- EINTRAG: ICTV Taxonomy history: Deltacoronavirus. In: Virus Taxonomy. International Committee on Taxonomy of Viruses, abgerufen am 22. Mai 2020 (englisch).
- PROPOSAL: Raoul J. de Groot and Alexander E. Gorbalenya: A new genus and three new species in the subfamily Coronavirinae. Proposal. In: International Committee on Taxonomy of Viruses (Hrsg.): Virus Taxonomy. History. Revision 2011. EC43, Sapporo September 2011, Proposal-Code 2010.023a-dV (englisch, ictvonline.org [PDF; 220 kB; abgerufen am 17. Juni 2020] Erstausgabe: EC 42, Paris 2010).
- ↑ a b c ICTV Code – The International Code of Virus Classification and Nomenclature. In: ICTV Homepage. International Committee on Taxonomy of Viruses, Oktober 2018, abgerufen am 16. Juni 2020 (englisch).
- ↑ a b Prof. Dr. John Ziebuhr: Medizinische Mikrobiologie und Infektiologie. Hrsg.: Sebastian Suerbaum, Gerd-Dieter Burchard, Stefan H. E. Kaufmann, Thomas F. Schulz. 8. Auflage. Springer, Berlin/Heidelberg 2016, ISBN 978-3-662-48677-1, S. 479 ff.
- ↑ Arnold S. Monto, Peter DeJonge, Amy P. Callear, Latifa A. Bazzi, Skylar Capriola, Ryan E. Malosh, Emily T. Martin, Joshua G. Petrie: Coronavirus occurrence and transmission over 8 years in the HIVE cohort of households in Michigan. In: Journal of Infectious Diseases. 4. April 2020, doi:10.1093/infdis/jiaa161.
- ↑ Common coronaviruses are highly seasonal, with most cases peaking in winter months. auf: ScienceDaily vom 7. April 2020, Quelle: University of Michigan
- ↑ Common Human Coronaviruses are Sharply Seasonal, Study Says. auf: Sci-News vom 8. April 2020.
- ↑ B.W. Neuman, M.J. Buchmeier: Coronaviruses. Nur Kapitel 1: Supramolecular Architecture of the Coronavirus Particle. In: Advances in Virus Research. 1. Auflage. Band 96. Elsevier Inc., 2016, ISBN 978-0-12-804736-1, ISSN 0065-3527, Abschnitt 2: Virion Structure and Durability, S. 4+5, doi:10.1016/bs.aivir.2016.08.005 (englisch, Volltext [PDF; 1,3 MB; abgerufen am 15. Juni 2020]).
- ↑ Yi Fan, Kai Zhao, Zheng-Li Shi and Peng Zhou: Bat Coronaviruses in China. In: Viruses. Band 11, Nr. 3. MDPI, 2. März 2019, S. 210, doi:10.3390/v11030210, PMID 30832341, PMC 6466186 (freier Volltext) – (englisch, Volltext [PDF; 0 kB; abgerufen am 16. Juni 2020]).
- ↑ NCBI Taxonomy browser: Pacific salmon nidovirus. In: NCBI-Homepage. National Center for Biotechnology Information (NCBI), abgerufen am 21. Mai 2020 (englisch).
- ↑ NCBI: Pacific salmon nidovirus (species)
- ↑ Gideon J Mordecai, Kristina M Miller, Emiliano Di Cicco, Angela D Schulze, Karia H Kaukinen, Tobi J Ming, Shaorong Li, Amy Tabata, Amy Teffer, David A Patterson, Hugh W Ferguson, Curtis A Suttle: Endangered wild salmon infected by newly discovered viruses. Version 1. In: eLife. eLife Sciences Publications Ltd., 3. September 2019, 47615, doi:10.7554/eLife.47615 (englisch, Volltext [PDF; 2,7 MB; abgerufen am 21. Mai 2020]): “The novel nidovirus, named Pacific salmon nidovirus (PsNV), is most closely related to the recently described Microhyla alphaletovirus 1 […].”
Information
Der Artikel Coronaviridae in der deutschen Wikipedia belegte im lokalen Ranking der Popularität folgende Plätze:
- 5. Platz im 2020-03-01
- 4. Platz im 2020-03-02
- 2. Platz im 2020-03-03
- 2. Platz im 2020-03-04
- 3. Platz im 2020-03-05
- 7. Platz im 2020-03-06
- 5. Platz im 2020-03-07
- 11. Platz im 2020-03-08
- 18. Platz im 2020-03-09
- 19. Platz im 2020-03-10
- 13. Platz im 2020-03-11
- 11. Platz im 2020-03-12
- 9. Platz im 2020-03-13
- 13. Platz im 2020-03-14
- 17. Platz im 2020-03-15
- 20. Platz im 2020-03-16
- 18. Platz im 2020-03-17
- 16. Platz im 2020-03-18
- 14. Platz im 2020-03-19
- 21. Platz im 2020-03-20
- 16. Platz im 2020-03-21
- 28. Platz im 2020-03-22
- 34. Platz im 2020-03-23
- 45. Platz im 2020-03-24
- 36. Platz im 2020-03-25
- 36. Platz im 2020-03-26
- 46. Platz im 2020-03-27
- 47. Platz im 2020-03-28
- 89. Platz im 2020-03-29
- 34. Platz im 2020-03-30
- 41. Platz im 2020-03-31
Über die Seite
Der präsentierte Inhalt des Wikipedia-Artikels wurde im 2021-06-12 basierend auf extrahiert https://de.wikipedia.org/?curid=4506862